
DNA甲基化电子书
了解DNA甲基化对于人类疾病和其他表型的研究至关重要。本电子书提供了分析DNA甲基化和执行表观基因组关联研究的注意事项,包括研究设计、样本类型和数据分析。
DNA methylation eBook
Your email address is never shared with third parties.
使用NGS技术进行亚硫酸氢盐测序,用于DNA甲基化测序分析
亚硫酸氢盐测序(BS-Seq)或全基因组亚硫酸氢盐测序(WGBS)是检测基因组DNA中甲基化胞嘧啶的成熟实验方案。在该方法中,先用亚硫酸氢钠处理基因组DNA,然后测序,从而得到单碱基分辨率的基因组甲基化胞嘧啶图谱。用亚硫酸氢钠处理DNA后,未甲基化的胞嘧啶脱氨形成尿嘧啶,并在测序后转化为胸腺嘧啶。同时,甲基化胞嘧啶能够抵抗脱氨基作用,因此会被识别为胞嘧啶。比较经处理与未经处理的序列即可确定甲基化胞嘧啶的位置1,2。


DNA甲基化电子书
了解DNA甲基化对于人类疾病和其他表型的研究至关重要。本电子书提供了分析DNA甲基化和执行表观基因组关联研究的注意事项,包括研究设计、样本类型和数据分析。
Your email address is never shared with third parties.

基因表达与调控电子书
基因调控是控制基因产物(包括mRNA和非编码RNA转录本)时空表达的生物学过程。了解现代转录组学和表观遗传学方法(包括BS-Seq和NGS技术)如何推动研究发现并更好地理解这一生物过程。
Your email address is never shared with third parties.

多组学的作用电子书
DNA通过遗传和表观遗传碱基储存分子信息,本质上具备多组学整合特性。将来自RNA-Seq的转录组信息与甲基化测序数据相结合,能够更好地了解细胞分化、机制和行为。深入了解并发掘多组学方法的强大作用。
Your email address is never shared with third parties.
简化代表性亚硫酸氢盐测序(RRBS-Seq)(单细胞简化代表性亚硫酸氢盐测序(scRRBS))是一种使用基因组DNA上的一种或多种限制酶来产生序列特异性片段化的实验方案。然后使用亚硫酸氢盐处理片段化的基因组DNA并测序。RRBS-Seq是研究特定感兴趣区域的选择方法。该方法对启动子区和重复序列区等甲基化水平较高的位置尤其有效3,4。
氧化亚硫酸氢盐测序(oxBS-Seq)可区分5mC与5hmC。使用oxBS时,5hmC会被氧化剂氧化为5-甲酰胞嘧啶(5fC),而5mC保持不变。氧化的5hmC经亚硫酸氢钠处理后脱氨基变为尿嘧啶,然后会被识别为胸腺嘧啶。对经过oxBS处理的DNA进行深度测序,并比对经处理和未经处理的序列,即能够以碱基分辨率确定5mC的位置5,6。
基于酶法片段化的全基因组亚硫酸氢盐测序(T-WGBS)是一种利用Tn5转座子和亚硫酸氢盐转化研究5mC的实验方案。与标准的WGBS方法相比,T-WGBS能够以更少的DNA(约20 ng)对材料进行测序。此外,Tn5转座子的优势还包括可在单个步骤中进行DNA片段化并连接测序接头。然后,用亚硫酸氢钠处理DNA并进行PCR扩增,最后测序7。
单细胞亚硫酸氢盐测序(scBS-Seq)是成熟的亚硫酸氢盐测序(BS-Seq)和亚硫酸氢盐处理后加接头标记(PBAT)方案的变型,改进后用于检测单细胞基因组DNA中甲基化的胞嘧啶。在该方法中,分离单个细胞后,基因组DNA经过亚硫酸氢钠处理,片段化DNA。转化后的DNA随后经过数次随机引导,并进行PCR扩增以供测序。深度测序以单核苷酸高分辨率呈现了单细胞的甲基化胞嘧啶8。
| 和缺点 | 亚硫酸氢盐测序的优势 |
亚硫酸氢盐测序的缺点 |
|---|---|---|
| BS-Seq |
|
|
| RRBS-Seq / scRRBS |
|
|
| oxBS-Seq |
|
|
| T-WGBS |
|
|
亚硫酸氢盐测序是检测DNA甲基化的常用方法,该方法利用亚硫酸氢盐将测序数据中未甲基化的C转化为T。然而,这种方法存在几个限制性。据估计,基因组中大约10%的CpG位点在亚硫酸氢盐转化后难以比对,并且存在可达到90%水平的DNA降解9。
作为亚硫酸氢盐测序的替代方案,因美纳5-碱基解决方案利用创新的测序化学技术和分析,可同时进行遗传变异和甲基化检测。因美纳5-碱基测序化学技术只需简单一步即可直接将5mC转化为T,不仅不会造成DNA损伤,还能保留文库复杂性。因美纳5-碱基解决方案可在单次检测中读取未经修饰的碱基(A、T、G、C)和5mC。
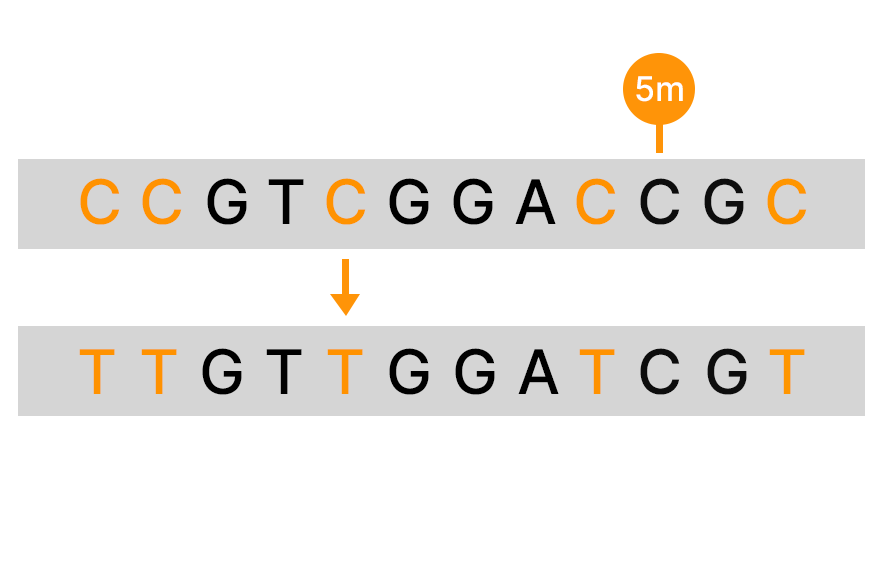
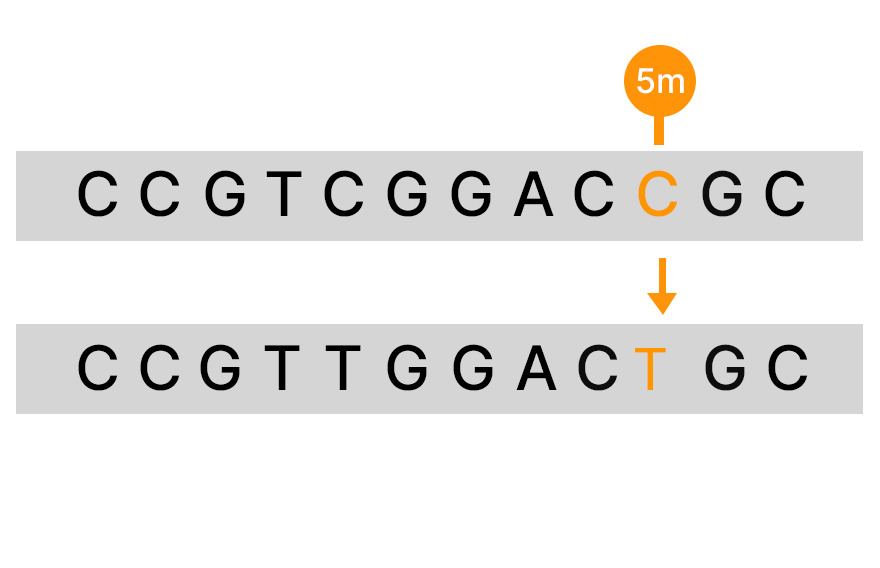
亚硫酸氢盐转化是亚硫酸氢钠选择性地将未甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶保持不转化的过程。亚硫酸氢盐处理通常与NGS联用,以实现全基因组DNA甲基化图谱分析。详细了解NGS的基本原理。
亚硫酸氢盐测序是一种常用于研究DNA甲基化的方法。在测序之前,通过用亚硫酸氢钠处理将未甲基化的胞嘧啶转换为尿嘧啶。然后比较亚硫酸氢盐处理和未处理样本的测序DNA,以确定甲基化和未甲基化胞嘧啶的位置。
用亚硫酸氢钠处理DNA可以使未甲基化的胞嘧啶通过化学脱氨基作用转化为尿嘧啶,而甲基化的胞嘧啶则不受此过程的影响。
亚硫酸氢盐转化过程和后续测序存在一些限制性,包括DNA降解、基因组复杂性降低以及比对过程的额外复杂度10。
靶向BS-Seq已被研究用于生物标记发现,并使用生物素标记的RNA探针等组分来捕获感兴趣的特定基因组片段。相反,WGBS可对整个基因组进行测序和分析11。
参考文献
与专家沟通,了解更多关于甲基化研究的解决方案。
Your email address is never shared with third parties.