Cancer Biomarker Detection

识别有意义的癌症生物标志物
与靶向疗法相关的癌症生物标志物数量在持续增加。目前,我们利用基于NGS的全景变异分析,通过一次活检、一次检测和一份报告就可以改善癌症患者的预后,推动精准医疗发展并改善患者的健康1-2。
为了更好地选择治疗方式,在评估癌症生物标志物时要考虑多个因素。
美国指南和高发肿瘤药物标签中的生物标志物1,3
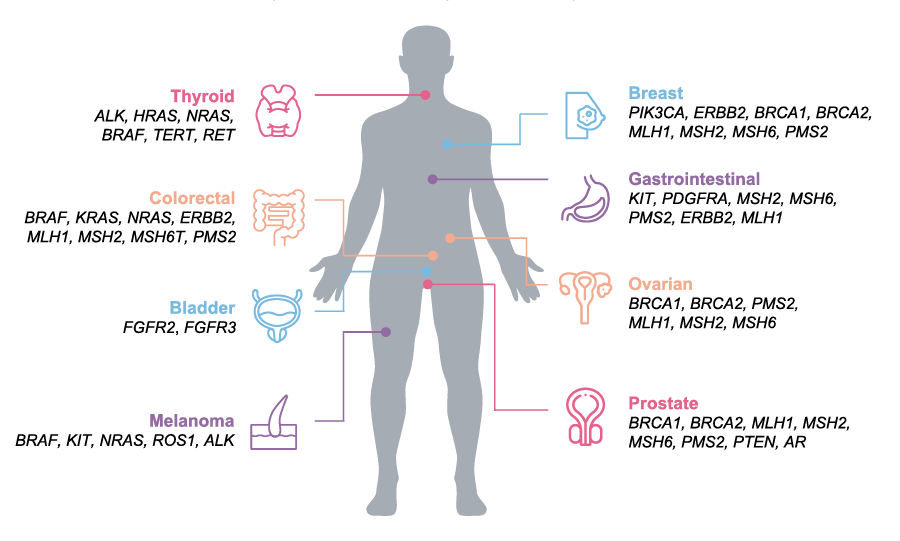
通过一次活检获得尽可能多的生物标志物数据
使用诸如FISH、PCR和IHC等技术进行常规检测无法评估所有相关癌症生物标志物,也无法保存珍贵的组织样本。连续进行多次单一生物标志物检测是非常常见的一种做法;不过,这也存在一些不足。首先,这些检测需要大量的活检样本,而获取这些样本并非总是可行。其次,这些单基因检测的内容有限,可能会漏检阳性生物标志物。
基于新一代测序(NGS)的生物标志物检测可以同时分析数百种具有临床意义的癌症生物标志物。这些额外的数据让医生有更多机会选择与患者相匹配的合适分子治疗方案。单次基于NGS的生物标志物检测可以替代多个单基因检测或小型热点panels.4-7。
无法进行组织活检时,液体活检的全景变异分析(CGP)可以提供有关肿瘤基因组组成的有用信息。这可以减少重新活检的需要,并在护理过程中节省宝贵的时间8。
同源重组修复缺陷(HRD)
同源重组修复缺陷(HRD)是一种用于描述HRR(同源重组修复)通路功能丧失的表型。当存在同源重组修复缺陷时,细胞无法修复双链DNA断裂,导致肿瘤形成。
HRD可通过“原因”(如BRCA突变)或“结果”(基因组瘢痕)进行评估。NGS技术可以同时评估致病基因和基因组瘢痕。
SNV、CNV和插入缺失
与癌症相关的基因突变有多种类型,如单核苷酸位点变异(SNV)、插入和缺失(indel)、拷贝数变异(CNV)等。
传统上,会采用单基因检测来分析这些特定的生物标志物,但随着每种癌症类型生物标志物数量的增加,越来越多的实验室开始采用NGS方法,这样可以在一次检测中分析众多基因和变异类型。
用于癌症生物标志物检测的CGP
全景变异分析(CGP)将数百种癌症相关生物标志物整合至一个检测中,无需进行连续检测。CGP能够在核苷酸水平检测生物标志物,并且通常包括所有主要的基因组变异类别(SNV、插入缺失、CNV、融合、剪接变异),以及大基因组特征(TMB 和 MSI),大幅提高了发现临床目标变异的能力。
深入了解CGP纳入CGP检测的癌症生物标志物
CGP可根据指南在单次检测中对数百个相关癌症生物标志物进行与肿瘤类型无关的检测,能够大幅节省样本、时间和成本。
| 泛癌生物标志物 NTRK1 NTRK2 NTRK3 MSI TMB RET BRAF | ||||||||
|---|---|---|---|---|---|---|---|---|
| 肺癌 | 黑色素瘤 | 结肠癌 | 卵巢癌 | 乳腺癌 | 胃癌 | 膀胱癌 | 肉瘤 | |
| AKT1 | BRAF | AKT1 | BRAF | AKT1 | BRAF | MSH5 | ALK | |
| ALK | CTNNB1 | BRAF | BRCA1 | AR | KIT | PMS2 | APC | |
| BRAF | GNA11 | HRAS | BRCA2 | BRCA1 | KRAS | TSC1 | BRAF | |
| DDR2 | GNAQ | KRAS | KRAS | BRCA2 | MET | CDK4 | ||
| EGFR | KIT | MET | PDGFRA | ERBB2 | MLH1 | CTNNB1 | ||
| ERBB2 | MAP2K1 | MLH1 | FOXL2 | FGFR1 | PDGFRA | ETV6 | ||
| FGFR1 | NF1 | MSH2 | TP53 | FGFR2 | TP53 | EWSR1 | ||
| FGFR3 | NRAS | MSH6 | PIK3CA | FOXO1 | ||||
| KRAS | PDGFRA | NRAS | PTEN | GLI1 | ||||
| MAP2K1 | PIK3CA | PIK3CA | KJT | |||||
| MET | PTEN | PMS2 | MDM2 | |||||
| NRAS | TP53 | PTEN | MYOD1 | |||||
| PIK3CA | SMAD4 | NAB2 | ||||||
| PTEN | TP53 | NF1 | ||||||
| RET | PAX3 | |||||||
| TP53 | PAX7 | |||||||
| PDGFRA | ||||||||
| PDGFRB | ||||||||
| SDHB | ||||||||
| SDHC | ||||||||
| SMARCB1 | ||||||||
| TFE3 | ||||||||
| WT1 | ||||||||
这里只展示了部分基因,不是详尽的清单。
特色在线讲座
威尔康奈尔医学院病理学实验室开始采用CGP
这些适用于因美纳试剂盒无人值守的自动化方法消除了开发您自己的方法的需求,帮助减少手动操作时间,减少错误。
甲状腺癌和NSCLC的新生物标志物
UCSD的Sandip Patel博士介绍了甲状腺癌和非小细胞肺癌(NSCLC)的生物标志物导向疗法,并讨论了这些适应症的关键新兴生物标志物及其支持性证据。
采用CGP实现广泛的肿瘤学生物标志物检测
医学博士Phil Febbo讨论了CGP的优势,以及它如何成为肿瘤学的全新标准治疗。
科学家讨论癌症生物标志物研究成果
识别对癌症免疫疗法产生反应和耐药性的相关生物标志物
Eliezer Van Allen博士重点介绍了NGS如何识别对癌症免疫疗法产生反应和耐药性的相关生物标志物,以及如何确定癌症如何对免疫检查点阻断产生耐药性。
准确识别癌症生物标志物
科学家们讨论了全面的基于NGS的panel对于发现生物标志物的意义。
NGS为生物标志物的发现提供了宝贵信息
医学博士Phil Febbo讨论了CGP的优势,以及它如何成为肿瘤学的全新标准治疗。
相关内容
肿瘤突变负荷
NGS方法可高效评估肿瘤突变负荷并鉴别新抗原。
ctDNA可作为非侵入性癌症生物标志物
NGS方法可高效评估肿瘤突变负荷并鉴别新抗原。
癌症表观遗传学
NGS和芯片技术可以检测到DNA甲基化模式的变化和其他调控癌症进展的表观遗传修饰。
参考文献
- US Food & Drug Administration. Hematology/Oncology (Cancer) Apporvals & Safety Notifications. US FDA website. Updated February 9, 2021. Accessed December 1, 2020.
- Singal G, Miller PG, Agarwala V, et al. Association of Patient Characteristics and Tumor Genomics With Clinical Outcomes Among Patients With Non-Small Cell Lung Cancer Using a Clinicogenomic Database. JAMA. 2019 Apr 9;321(14):1391-1399. doi: 10.1001/jama.2019.3241.
- PierianDx. Next Generation Knowledge. PierianDx - Genomic Knowledgebase for Clinical Next Generation Knowledge. Accessed March 1, 2020.
- Malone ER, Oliva M, Sabatini PJB, Stockley TL, Siu LL. Molecular profiling for precision cancer therapies. Genome Med. 2020 Jan 14;12(1):8. doi: 10.1186/s13073-019-0703-1.
- Reitsma M, Fox J, Borre PV, et al. Effect of a Collaboration Between a Health Plan, Oncology Practice, and Comprehensive Genomic Profiling Company from the Payer Perspective. J Manag Care Spec Pharm. 2019 May;25(5):601-611. doi: 10.18553/jmcp.2019.18309.
- Kopetz S, Mills Shaw KR, Lee JJ, et al. Use of a Targeted Exome Next-Generation Sequencing Panel Offers Therapeutic Opportunity and Clinical Benefit in a Subset of Patients With Advanced Cancers. JCO Precis Oncol. 2019 Mar 8;3:PO.18.00213. doi: 10.1200/PO.18.00213.
- Drilon A, Wang L, Arcila ME, et al. Broad, hybrid capture-based next-generation sequencing identifies actionable genomic alterations in lung adenocarcinomas otherwise negative for such alterations by other genomic testing approaches. Clin Cancer Res. 2015 Aug 15;21(16):3631-9. doi: 10.1158/1078-0432.CCR-14-2683.
- Rolfo C, Mack P, Scagliotti G, et al. Liquid biopsy for advanced NSCLC: a consensus statement from the international association for the study of lung cancer. J Thorac Oncol. 2021 Oct;16, (10):1647-62. doi: 10.1016.