NGS术语指南
规划测序项目时可使用我们的新一代测序术语表来阐明关键术语和重要概念。
NGS术语表
短序列特异性寡核苷酸,在NGS文库制备时会连接到测序文库中每个DNA片段的5’和3’末端。这些接头与Illumina流动槽表面的短序列互补。
将测序read与参考基因组进行匹配的过程。
成对的标签,每个i5标签将与矩阵中的i7标签配对,形成唯一标签对,但不是唯一的单侧标签。
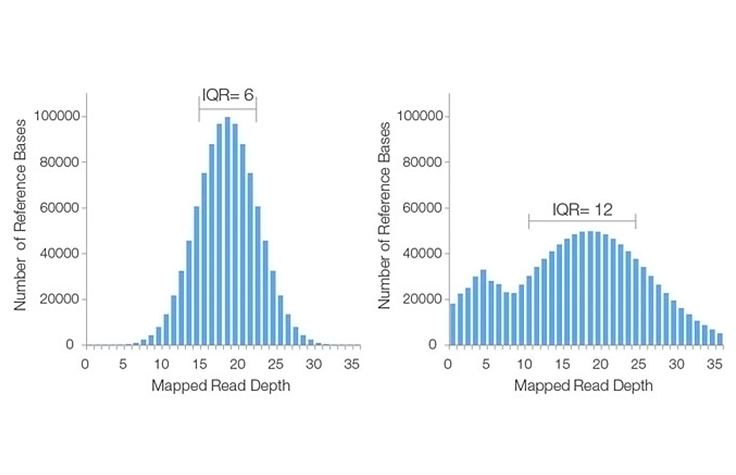
比对到或“覆盖”已知参考碱基序列的测序碱基平均数量。例如,以30倍覆盖度测序的全基因组意味着基因组中的每个碱基平均被测序30次。覆盖度水平越高,碱基检出的可信度越高。
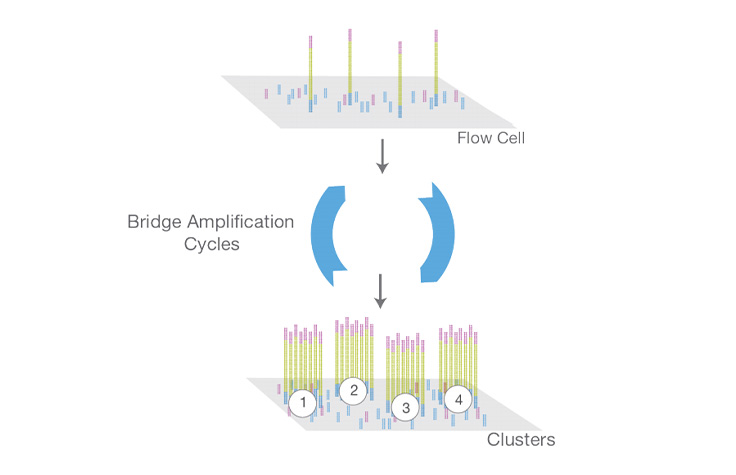
了解更多一种载破片或其他表面具有一条、两条或八条物理隔离道的实体,它是一种NGS仪器耗材。测序模板固定于流动槽表面,其被设计为以便于获得酶的方式呈递DNA,同时能确保表面结合模板的高稳定性和荧光标记核苷酸的低非特异性结合。固相扩增(簇生成)可在附近为每个单模板分子的生成多达1000个相同的拷贝。密度可达到每平方厘米一千万个簇。

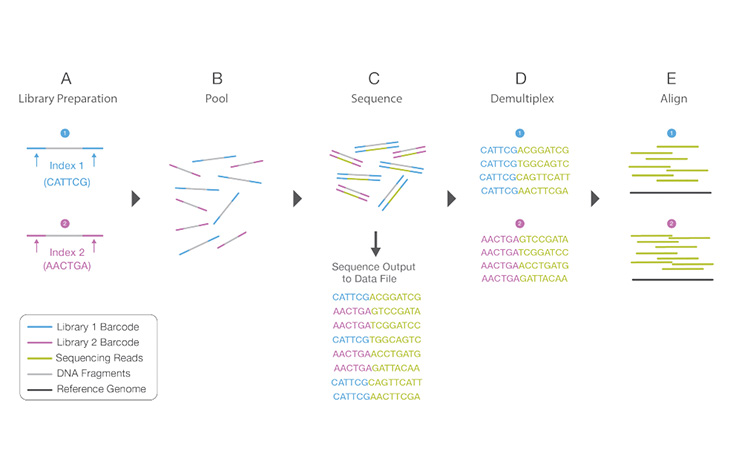
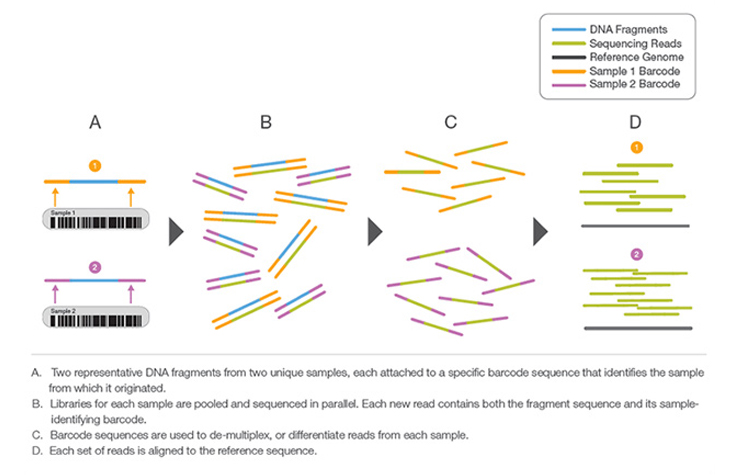
一段唯一的短DNA序列,在文库制备时加到每个DNA片段中。这段唯一的序列可以让多个文库混合在一起然后同时进行测序。在最终的数据分析之前,通过计算的方法,根据条形码对混合文库的测序read进行识别与分类。处理小型基因组或靶定感兴趣的基因组区域时,文库多重分析是一种有用的技术。利用条形码进行多重分析可大大增加每次运行分析的样本数量,而不会显著增加运行成本或运行时间。
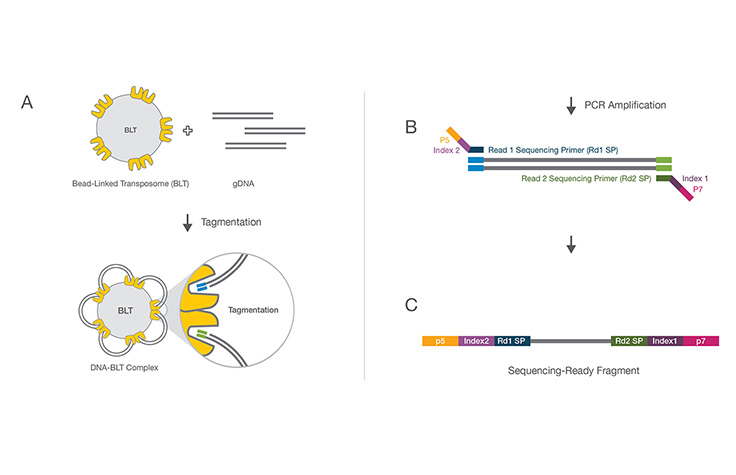
在文库制备过程中,样本DNA被片段化为特定大小(通常为200–500 bp,但可能会更大),然后连接或“插入”到两个寡核苷酸接头之间。原来的样本DNA片段也被称为“插入片段”。
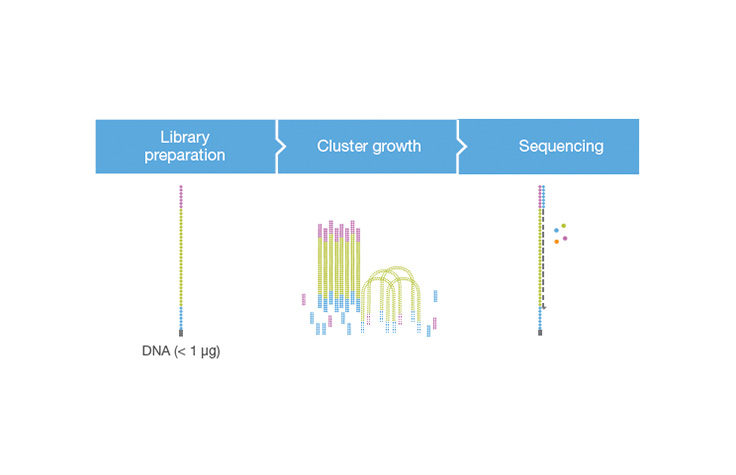
一种分子生物学实验方案,可以将基因组DNA样本(或cDNA样本)转换为测序文库,然后可在NGS仪器上进行测序。文库制备的第一步是DNA样本的随机片段化,然后将每个DNA片段连接上5ʹ和3′接头。或者,采用“酶法片段化”将片段化和连接反应合并成一个步骤,可极大地提高文库制备过程的效率。
了解更多新一代测序的另一个名字。
在文库制备时,向每个DNA片段添加唯一的短DNA序列(或“标签”)的过程。这段唯一的序列可以让多个文库混合在一起然后同时进行测序。在最终数据分析之前,通过计算的方法对混合文库的测序read进行识别与分类。处理小型基因组或靶定感兴趣的基因组区域时,文库多重分析是一种有用的技术。多重分析可大大增加每次运行所分析的样本数量,而不会显著增加运行成本或运行时间。
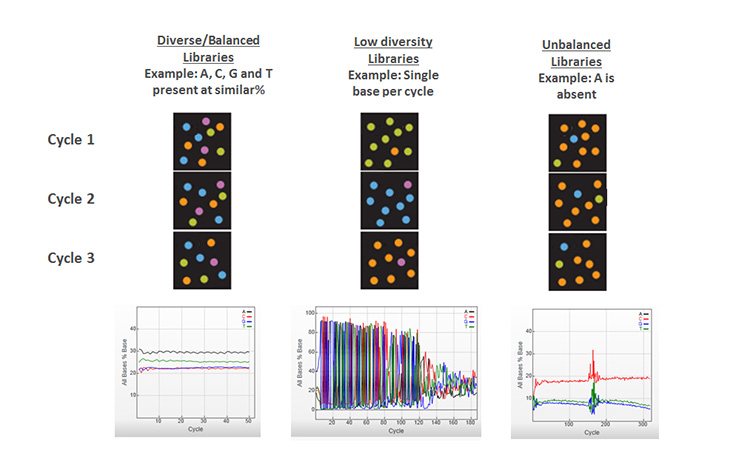
了解更多在测序文库的所有DNA片段的每个碱基位置上具有相同比例的A、C、G和T核苷酸。色彩平衡有助于在Illumina测序系统上进行有效的图像分析。因此,大部分Illumina文库制备工作流程包含一个随机片段化的步骤,该步骤会在文库的每个碱基位置生成必要的序列多样性。
阅读公告一段短的DNA或RNA序列。
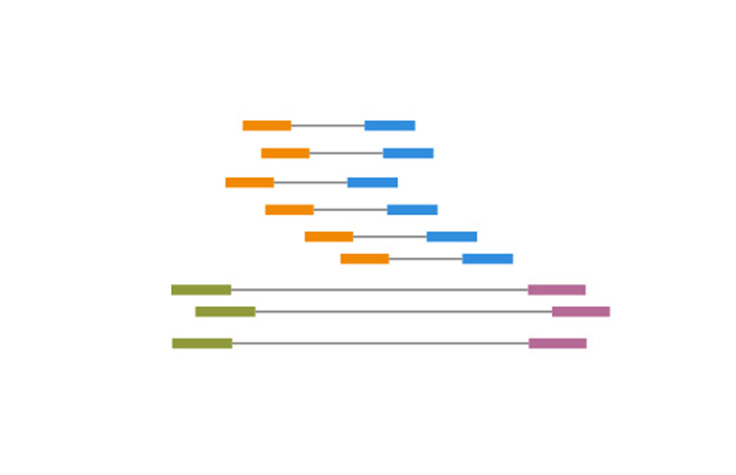
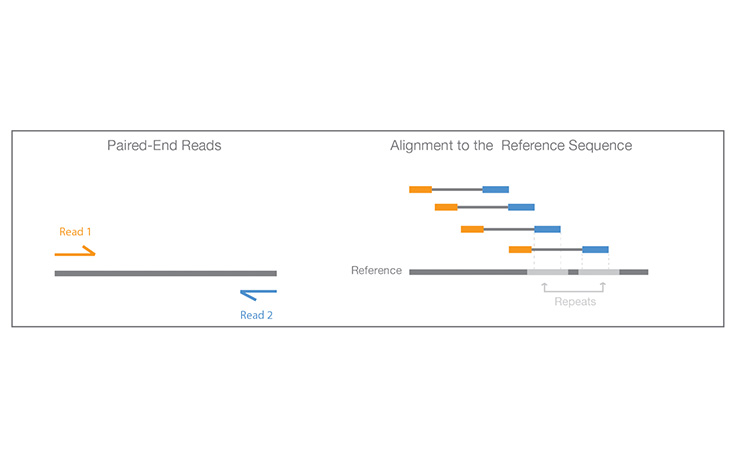
在同一运行中,从DNA片段的两端进行测序的过程。
了解更多NGS中的一种度量指标,可预测或估算碱基检出的错误率。质量分值(Q值)是一种表示极小错误率的简洁方式。高Q值表明碱基检出结果可靠,错误的可能性较低。
了解更多参考基因组是一种已完全测序并组装好的基因组,可发挥scaffold的作用,以比对和比较新的测序read。一般而言,作为数据分析的第一步,从测序运行生成的read将与参考基因组进行比对。参考基因组的示例包括hg19和hg38。
指检测给定样本中特定变异的能力。等位基因频率越低,则检测其所需的灵敏度越高。相较于毛细管电泳,新一代测序拥有更高的灵敏度,能检测罕见突变。
一段与接头序列相邻的PCR引物,可指示出测序read的起始位点。测序过程中,引物会退火到模板链的测序接头上。DNA聚合酶会结合到该位点,将互补的核苷酸碱基逐个地连接到延伸的相反链中。
测序文库中每个DNA片段对应的A、T、C、G碱基数据串。在Illumina的技术流程中,文库测序时,每个DNA片段会在流动槽表面生成一个簇,每个簇会生成一个测序read。(例如,流动槽上100万个簇将产生100万条单端read和200万条双端read。)读长范围在25 bp到300 bp之间或更高,具体取决于应用的需求。
底物上荧光基团的亮度。边合成边测序通过测定每个循环的信号强度来直接进行碱基检出。
Illumina支持多种标签方法,包括单标签和双标签。采用单标签的方式,最多可使用48个唯一的6碱基标签生成48个具有唯一标记的文库。采用双标签的方式,最多可以组合使用24个唯一的8碱基标签1序列和16个唯一的8碱基标签2序列,生成多达384个具有唯一标记的文库。
靶向重测序实验中所有目标区域的总体大小。目标大小因预设计panel或定制panel的不同而有所不同。(例如,一个panel有22个目标区域,每个目标区域为100 kb,则总目标大小为2200 kb。)
新一代测序仪器生成的数据量。通常以兆碱基(Mb)或千兆碱基(Gb)定义。
1兆碱基 = 1,000,000碱基。
1千兆碱基 = 1,000,000,000碱基。
1兆碱基 = 1,000,000碱基。
1千兆碱基 = 1,000,000,000碱基。